Lo Staphylococcus aureus è un patogeno Gram-positivo responsabile di infezioni sanitarie e di comunità (polmonite, osteomielite, endocardite, shock settico, ecc.). S. aureus è in grado di sviluppare resistenza contro molte classi di antibiotici e quindi sfuggire alla maggior parte delle opzioni terapeutiche. Le strategie sviluppate per combattere le infezioni stafilococciche refrattarie includono la terapia di combinazione e l’uso di antibiotici di ultima istanza. Tuttavia, la resistenza a questi farmaci sta iniziando a emergere a livello globale, esaurendo le opzioni per trattare con successo le infezioni da stafilococco.
S. aureus fa affidamento sul ferro per l’invasione dell’ospite e instaurare l’infezione. Il batterio possiede almeno tre meccanismi per approvvigionarsi di ferro dall’ospite: siderofori, recettori della transferrina/lattoferrina ed emofori. Quello preferito è la cattura dell’eme dall’emoglobina ospite (Hb) da parte del sistema di emofori Iron-regulated surface determinant (Isd), composto da nove proteine che legano l’Hb, estraggono l’eme, lo trasferiscono attraverso la parete cellulare e la membrana e lo ossidano per rilasciare il ferro (Figura 1).
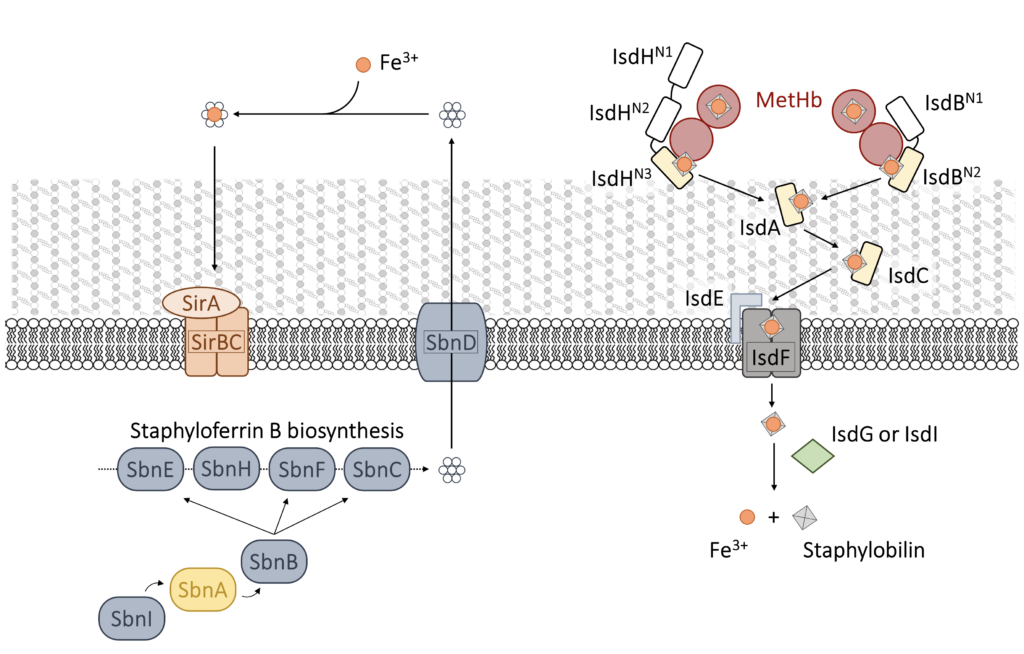
Il primo passaggio obbligato nella cattura dell’eme viene eseguito da due proteine isofunzionali ancorate alla parete cellulare: IsdB e IsdH. In passato, IsdH è stato studiato in modo più dettagliato, ma IsdB è probabilmente il principale attore nell’acquisizione dell’eme durante l’infezione ed è stato identificato come un fattore di virulenza di S. aureus. Queste proteine sfruttano i domini NEAT (NeAr iron Transporter) per legare selettivamente l’Hb umana ed estrarre l’eme che viene poi trasferito ad altre proteine del sistema. Sia IsdB che IsdH legano Hb con un dominio NEAT attraverso un’interazione specifica che coinvolge residui conservati di Tyr e catturano l’eme con un altro dominio NEAT (Figura 2).

Studi strutturali hanno posto le basi per identificare le superfici di interazione proteina-proteina (PPI) e hanno spinto il nostro gruppo ad iniziare uno studio in silico e in vitro con l’obiettivo di identificare potenziali inibitori in grado di influenzare la prima fase del riconoscimento emoforo-Hb e quindi, la conseguente estrazione dell’eme.
Abbiamo già identificato mediante screening virtuale molecole che prendono di mira l’interazione IsdB-Hb e attualmente le stiamo testando per la loro capacità di destabilizzare il complesso mediante un test ELISA, SPR e STD-NMR.
Oltre al sistema Isd, durante l’infezione viene indotto un meccanismo aggiuntivo per l’acquisizione di ferro, la produzione di siderofori (stafiloferrina A – SA, e stafiloferrina B – SB) (Figura 1). Il primo stadio obbligato della biosintesi di SB è effettuato da SbnA, un enzima PLP-dipendente che catalizza la beta-sostituzione di O-fosfoserina (OPS) con glutammato. La nostra esperienza nell’enzimologia degli enzimi PLP-dipendenti e nel loro sfruttamento come potenziali bersagli per antibatterici innovativi può essere applicata alla ricerca di inibitori di SbnA.
Attaccare due diversi meccanismi di acquisizione del ferro aumenterà la possibilità di sviluppare nuovi antibatterici che potrebbero essere utilizzati anche in combinazione.
